转录因子是调控基因表达的重要蛋白质,它能够结合到基因的特定区域并控制基因的转录过程。预测转录因子结合位点的研究可以帮助人们更好地理解转录因子的功能和基因调控网络的复杂性,同时也可以揭示基因表达的调控机制。预测转录因子结合位点在研究疾病的发病机理方面也具有重要意义,它可以帮助人们发现和解释与疾病相关的基因变异和突变,进而探索潜在的治疗策略。此外,在药物研发中,预测转录因子结合位点可以帮助人们设计更加精准的靶向药物,从而提高药物的疗效并降低副作用。随着高通量测序技术的不断发展,新的转录因子和结合位点不断被发现,因此开发高效、准确的人工智能方法来预测转录因子结合位点具有重要意义。这些方法可以为生物医学领域的研究提供更有价值的信息,进一步推动基因调控网络的研究和生命科学的发展。

近日,金沙集团1862cc信息科学技术学院2019级本科生余雨桐在牛津大学出版社出版的生物信息学顶级期刊Briefings in Bioinformatics (IF=13.994) 发表了题为“Cooperation of local features and global representations by a dual-branch network for transcription factor binding sites prediction”的高水平论文,报道了构建预测转录因子结合位点的人工智能算法模型—DSAC。该模型表现出较强的特征学习能力以及优异的泛化性。
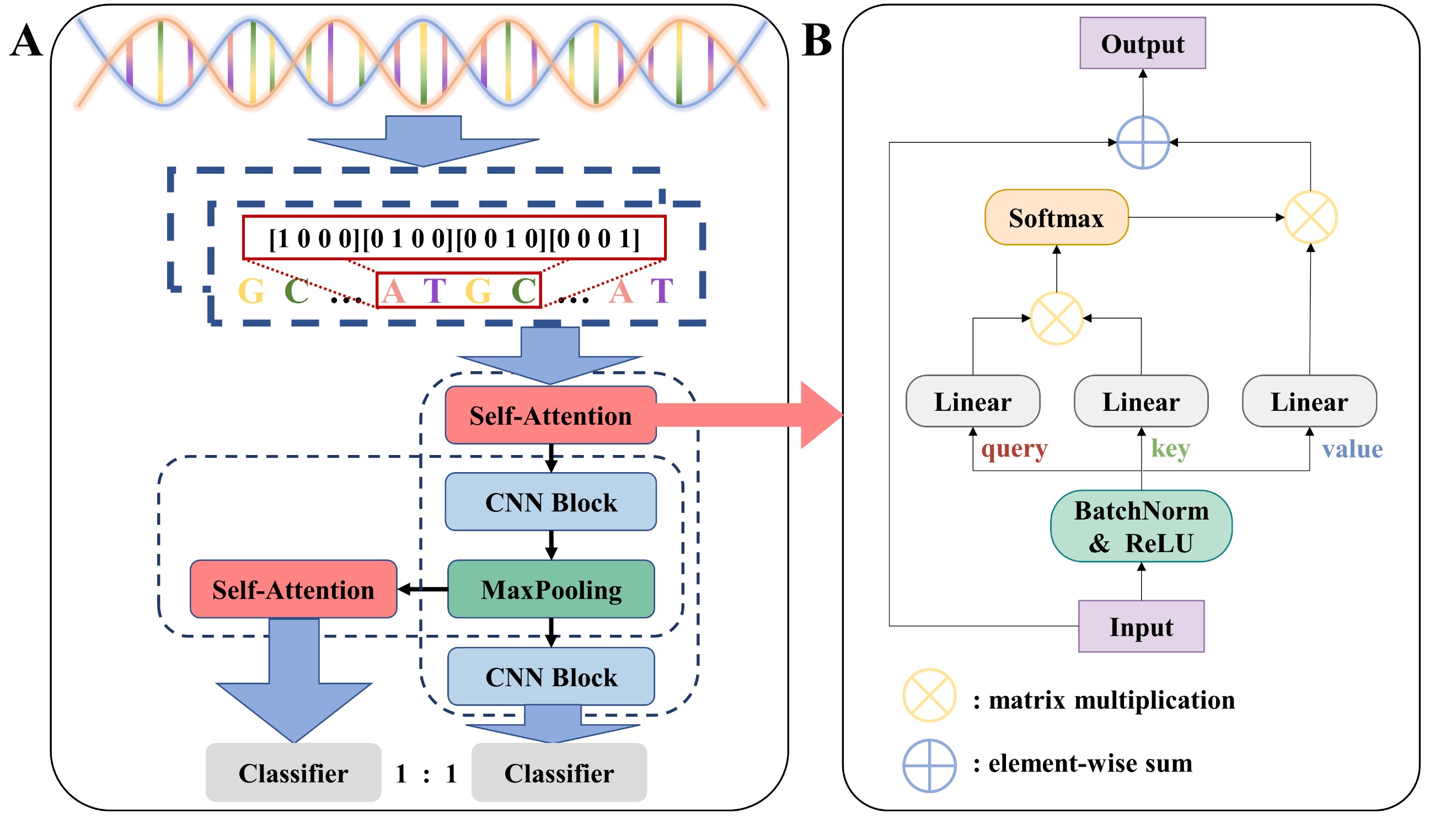
本研究首次提出一种新颖的双分支结构,将卷积神经网络与自注意力机制结合到转录因子结合位点的预测。自注意力机制能够加权处理输入序列中不同位置的信息,提高长距离依赖关系的建模能力,有助于检索具有序列特征的转录因子结合位点。卷积神经网络能够对输入数据进行局部特征提取,并且具有平移不变性,对于输入数据的轻微变化能够保持相对稳定的输出结果,从而在基因组序列数据的噪声和变异处理方面具有高效性。本研究提出的模型DSAC将两者有效的结合在双分支框架中,并且采用1:1的权重比,结合全局信息和局部特征,使得其在与其它深度学习的先进模型对比中突显出更强的特征提取能力与更优异的泛化性,以此来达到更好的预测效果。本文提出的DSAC深度学习模型能够成为生物信息学的有效工具,为揭示基因调控机制、研究疾病发生机制以及处理大规模基因组数据等方面做出贡献。
该研究成果的第一作者是信息学院2019级本科生余雨桐,于彬教授为论文的通讯作者,金沙集团1862cc为第一完成单位。
文章链接:https://doi.org/10.1093/bib/bbad036
余雨桐,信息学院计算机192班本科生,在校期间参与全国大学生数学竞赛,软件测试大赛,蓝桥杯等比赛,获得多个奖项。大三上学期进入于彬教授领导的人工智能与生物医学大数据研究团队,学习深度学习、生物信息学的有关理论与方法,并开展关于转录因子结合位点预测的研究工作。






